Un diario, dove annoto tutto ciò che più mi colpisce. Il mio blocco per gli appunti, il mio mondo.
giovedì 23 ottobre 2025
sabato 9 agosto 2025
Carte cosmiche di 6000 anni fa.
venerdì 18 luglio 2025
DNA - genoma umano -
Pensa un po': quello che credi di sapere sul tuo DNA è completamente sbagliato.
Ti hanno sempre detto che il DNA è come un manuale di istruzioni fisso, scritto una volta per tutte alla nascita. Roba da fantascienza quello che sto per dirti.
Quasi metà del tuo DNA, per la precisione il 45%, è fatto di 'geni saltatori'. Sono antichi virus che milioni di anni fa hanno invaso il genoma dei nostri antenati e ci sono rimasti. Per sempre.
Ecco il bello: questi 'invasori' non se ne stanno fermi. Funzionano come un copia-incolla impazzito, spostandosi e duplicandosi nel nostro DNA per tutta la vita. Praticamente il nostro genoma è un campo di battaglia dove antichi virus continuano a riscrivere il codice genetico.
In pratica, non siamo solo umani. Siamo degli ibridi: parte umano, parte virus antico. Il nostro DNA è un museo vivente di invasioni virali che risalgono all'alba della vita.
Altro che manuale fisso: il nostro genoma è un libro che si riscrive da solo, pagina dopo pagina, con l'aiuto di questi antichi squatter microscopici.
Incredibile pensare che portiamo dentro di noi la storia di battaglie evolutive combattute milioni di anni fa.
https://www.facebook.com/photo/?fbid=1177649547725724&set=a.447499420740744
Gli architetti del genoma arrivano dal passato. - Francesco Centorrino
Progetto Genoma Umano.
Il 14 aprile 2003 è stato portato a termine il Progetto Genoma Umano: finalmente si disponeva del completo sequenziamento del DNA della nostra specie. Alla gioia dell’impresa eroica è tuttavia susseguita subito l’incomprensione: la parte codificante dei geni (gli esoni), rappresenta infatti soltanto una piccola percentuale del nostro DNA.
I trasposoni.
In cosa consiste il resto del genoma? La maggior parte dei nostri cromosomi è costituita da sequenze ripetitive disperse: i trasposoni. I nostri cromosomi sono un oceano di trasposoni dal quale emergono di tanto in tanto gli esoni.
I trasposoni, anche chiamati elementi genetici mobili, elementi trasponibili o geni saltatori, sono dei frammenti di DNA capaci di replicarsi e di posizionarsi in regioni diverse dei cromosomi. Questo è un meccanismo molecolare spesso codificato dall’elemento stesso.
Qual è il loro ruolo?
Sembra che la storia dei trasposoni sia molto antica: essi hanno oltrepassato le barriere inter-specie acquisendo delle funzioni ritenute essenziali. Tuttavia, succede anche che i trasposoni possano seminare il panico nelle nostre cellule ed innescare processi patologici, come quelli cancerosi.
“Taglia – incolla” o “incolla – taglia”?
A seconda dei loro meccanismi, si distinguono due grandi famiglie di trasposoni: “taglia – incolla” o “incolla – taglia”. I primi prevedono l’escissione sotto forma di DNA, accompagnata dall’integrazione della parte escissa in un’altra parte del genoma. Essi rappresentano la famiglia dei trasposoni a DNA, spesso chiamati solo trasposoni (Figura 1).
L’altra famiglia, detta “incolla – taglia”, è invece la più comune negli esseri umani. In questo caso l’elemento è dapprima trascritto in RNA, convertito in DNA ed integrato in un’altra parte del genoma. In questo caso si parla di retrotrasposoni. L’enzima chiave di questo processo è la trascrittasi inversa, che ricopia l’RNA sotto forma di DNA. Esso è presente anche nei retrovirus, come HIV, i quali ne rappresentano i probabili antenati. Del resto, la distinzione tra retrotrasposoni e retrovirus è incerta.
Alcuni retrotrasposoni sono diventati indipendenti dall’ospite originale dopo aver acquisito un gene codificante per proteine dell’involucro, divenendo retrovirus. Al contrario, certi retrovirus hanno infettato la linea germinale del loro ospite (le cellule sessuali) diventando così dei retrotrasposoni, trasmessi da una generazione ad un’altra.
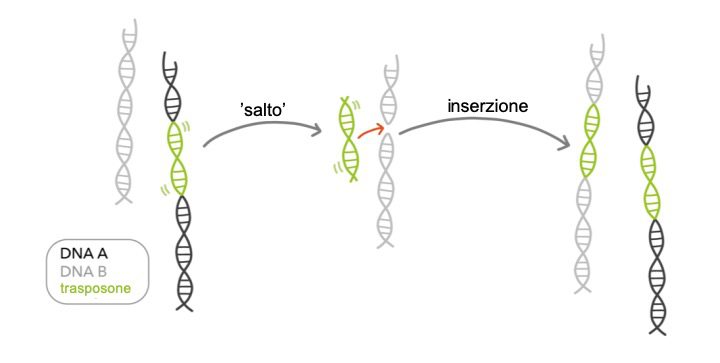
I retrotrasposoni della linea L1
Solo una sottofamiglia di retrotrasposoni è sempre attiva nel nostro genoma: i retrotrasposoni della linea 1 o L1, di origine non virale. Essi assistono la replicazione (retrotrasposizione) di elementi trasponibili non codificanti per una trascrittasi inversa, come le sequenze Alu o SVA. Tuttavia, tra le migliaia di copie di elementi L1 del nostro genoma, solo un centinaio sono in grado di generare nuove copie. Gli elementi L1 sono presenti solo negli umani; i nostri cugini, scimpanzè e gorilla, possiedono infatti altri elementi trasponibili attivi.
In seguito al Progetto Genoma Umano, è stato sequenziato il genoma di migliaia d’individui diversi nel mondo. Seppur simili, essi differiscono tuttavia in modo più o meno importante. Le variazioni genetiche riscontrate inoltre non sono soltanto semplici modificazioni di nucleotidi, ma anche cambiamenti più grandi, come delezioni o inserzioni, in particolare proprio di elementi trasponibili.
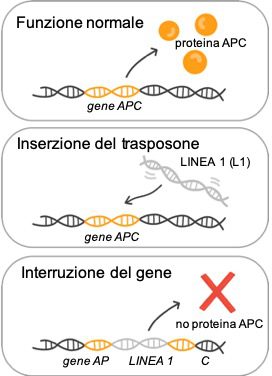
Le conseguenze dei trasposoni possono essere complesse: si potrebbero verificare ad esempio modificazioni dei livelli d’espressione del gene, perturbazioni del profilo spazio-temporale della sua espressione, persino creazioni di nuove forme dello stesso gene (Figura 2).
Alla fine degli anni ’80, Haig Kazazian, studiò le mutazioni responsabili dell’emofilia. Kazazian scoprì delle inserzioni dell’elemento L1 nella regione codificante del gene del fattore VIII (implicato nella coagulazione), nei pazienti affetti da emofilia. Quest’osservazione è stata straordinaria per diversi motivi: ha infatti dimostrato non solo che esistevano degli elementi trasponibili attivi negli esseri umani, ma anche che potevano essere la causa di malattie genetiche.
Oggi sappiamo che inserzioni ereditabili possono prodursi nelle cellule sessuali, spermatozoi e ovuli, dei genitori. Fortunatamente, queste nuove inserzioni non provocano necessariamente malattie genetiche.
I mosaici genetici.
Il lavoro di Kazazian e, più recentemente, il lavoro del team di Jose-Luis Garcia-Perez dell’Università di Edimburgo, mostrano che il processo di retrotrasposizione potrebbe avvenire nel primo stadio dello sviluppo embrionale.
In questo stadio infatti alcune cellule dell’organismo o della placenta sono soggette ad inserzione ed altre no, facendo sì che molti di noi risultino chimere genetiche. Nella maggior parte dei tessuti adulti, i retrotrasposoni L1 sono repressi da numerosi meccanismi cellulari che bloccano la loro espressione e replicazione.
I lavori di Alysson Muotri e Fred Gage, dell’Istituto Salk a San Diego, hanno mostrato che i retrotrasposoni sono reattivi nel cervello degli esseri umani e dei roditori. Le domande restano comunque tante: è un processo normale, oppure importante per le funzioni cerebrali o la complessità del nostro cervello? Una maggiore attività dei retrotrasposoni potrebbe dare origine a delle malattie neurologiche o neurodegenerative, come la schizofrenia o il morbo di Parkinson?
Il laboratorio di Fred Gage ha rivelato che nel topo, l’accumulazione di nuove copie dell’elemento L1 potrebbe essere influenzata dalle prime esperienze di vita, collegando così ambiente e alterazioni genetiche nel cervello. I retrotrasposoni L1 sono attivi anche nei tumori umani. Il gruppo di Burns, dell’università John Hopkins, ha individuato il meccanismo di retrotrasposizione in circa la metà dei tumori epiteliali. Numerosi laboratori nel mondo hanno scoperto nuove inserzioni di L1 nei tumori, e la loro assenza nei tessuti normali circostanti.
Recentemente, un vasto studio del Consorzio Internazionale della Genomica del Cancro, ha provato che la riattivazione dei retrotrasposoni può condurre a un largo ventaglio d’alterazioni cromosomiche. In particolare, nel cancro dell’apparato digerente (cavo orale, esofago, stomaco e colon) o del polmone. I genetisti hanno identificato migliaia d’inserzioni, ma anche inversioni, traslocazioni o grandi delezioni di milioni di paia di basi, associate alle inserzioni.
Trasposoni ed epigenetica.
E cosa succede a livello epigenetico? Le modificazioni chimiche del DNA e degli istoni, le proteine attorno alle quali il DNA si avvolge, giocano un ruolo importante nella regolazione genica, e nella repressione dei retrotrasposoni. Questo profilo epigenetico è profondamente alterato nelle cellule cancerose. Inoltre, si è pensato per molto tempo che queste alterazioni erano sufficienti per riattivare i retrotrasposoni L1.
La verifica di quest’ipotesi è stata difficile per due ragioni: la sequenza dei retrotrasposoni è quasi identica e la loro posizione nel genoma varia enormemente. In queste condizioni com’è possibile sapere se nelle cellule tumorali molti elementi L1 sono riattivati, o se solamente un piccolo numero di copie viene espressa?
Alcuni ricercatori hanno utilizzato l’associazione di più approcci innovativi di sequenziamento e bioinformatica per arrivare a formulare una risposta: solo poche copie di L1 sono attive in un tipo cellulare o in un dato tessuto. Alcune di queste copie sono polimorfiche nella popolazione, sono cioè presenti solo in certi individui, possono rappresentare dei fattori di predisposizione al cancro nel tessuto dove sono attivi. In altre parole, non siamo tutti uguali per i retrotrasposoni e i loro effetti potenzialmente mutageni.
Inserzioni aleatorie… o quasi.
Resta una domanda essenziale: gli elementi trasponibili s’inseriscono in modo totalmente aleatorio nel genoma, oppure prendono di mira delle regioni particolari? Lo studio di organismi modelli e dei retrovirus ha messo in luce la diversità delle strategie adattate: alcuni elementi trasponibili privilegiano le regioni più accessibili dei cromosomi, altri le regioni particolarmente compatte.
Alcuni preferiscono i geni, altri li evitano. Tuttavia, due studi mostrano che seppur non si inseriscano in modo omogeneo nel nostro DNA, non sono affatto influenzati dallo stato di condensazione della cromatina o dalla presenza di geni.
Il principale fattore che guida la loro integrazione è la presenza di un motivo molto corto (una sequenza di basi del DNA) riconosciuto da degli enzimi codificati dall’elemento L1 che guidano la retrotrasposizione. Questo motivo non è distribuito nello stesso modo in tutte le regioni cromosomiche, né tra i due filamenti della doppia elica di DNA. Perché questi risultati sono importanti? Paragonando le regioni dove un elemento L1 si può inserire e quelle dove lo ritroviamo nel corso dell’evoluzione di una popolazione, possiamo riuscire a capire in quali regioni l’inserimento di un elemento trasponibile è dannoso. Viceversa, l’arricchimento d’inserzioni in una regione, per esempio in un tumore, può mettere in luce i fattori che contribuiscono alla crescita tumorale.
Il caso della sindrome di Aicardi-Goutières.
Per quanto riguarda gli effetti mutageni dei retrotrasposoni attraverso la loro inserzione nel genoma, lo studio della sindrome di Aicardi-Goutières, dal gruppo di Daniel Stetson, all’università di Washington ha aperto nuove prospettive.
I pazienti colpiti da questa malattia genetica rara presentano dei sintomi infiammatori, disordini neuorologici e dello sviluppo importanti, soprattutto nel cervello. Questo insieme di sintomi ricordano quelli di un’infezione virale congenitale, ma nessun virus conosciuto è stato identificato. La risposta infiammatoria sembra essere iniziata dalla produzione di DNA complementare del retrotrasposone L1 ed il suo accumulo nel citoplasma delle cellule. Si ignora come queste molecole di DNA possano lasciare il nucleo dove la retrotrasposizione avviene normalmente.
Sorprendentemente, in caso di senescenza cellulare, in cui si assiste alla perdita della facoltà proliferativa della cellula a causa del suo invecchiamento, si manifesta un fenomeno simile. La cromatina e gli altri marcatori epigenetici sono profondamente rimodellati, i retrotrasposoni L1 sono riattivati, senza che si notino fenomeni d’inserzione: il loro DNA complementare si accumula nel citoplasma e attiva la risposta infiammatoria legata all’interferone.
Questa risposta infiammatoria, all’origine di numerose patologie associate all’invecchiamento, è abolita quando le cellule, o i topi, sono trattati con inibitori della trascrittasi inversa, utilizzati contro l’HIV. Questa scoperta, fatta dal gruppo di John Sedivy dell’università di Brown, suggerisce che i retrotrasposoni sarebbero direttamente implicati nell’invecchiamento, e che sarebbe possibile limitare il loro effetto deleterio attraverso degli approcci farmacologici.
Nuovi sistemi di connessioni geniche
Le sequenze regolatrici dei trasposti possono agire anche a distanza. Esse sono ridistribuite nel genoma in seguito alla migrazione degli elementi trasponibili con degli effetti diversi a seconda che si inseriscano vicino o all’interno di un gene.
Barbara McClintock, premio nobel della medicina nel 1983 per la scoperta dei trasposoni, suggerì per prima che questi fossero in grado di controllare i geni. Mentre, Roy Britten e Eric Davidon negli anni 1970, immaginarono che essi potessero essere distribuiti e creare nuovi moduli genetici, permettendo un’evoluzione ed un adattamento rapido ad un ambiente mutevole.
Gli esempi nel mondo vegetale e animale sono numerosi. Per esempio, l’inserzione dei retrotrasposoni a monte di un gene implicato nella regolazione della biosintesi delle antocianine, ha portato allo sviluppo delle arance sanguigne.
Oppure, all’inizio del XIX secolo, l’inserzione di un elemento trasponibile in un gene legato alla pigmentazione delle falene, le ha rese più scure. Quest’adattamento fù essenziale, perché permise alle falene di difendersi dagli uccelli, loro predatori. Infatti, in piena rivoluzione industriale, i tronchi degli alberi erano più scuri a causa dell’inquinamento atmosferico.
Il gruppo di Cédric Feschotte, dell’università di Utah, ha identificato negli esseri umani una famiglia di retrovirus endogeni (MER41), che ha invaso il nostro genoma tra 45 a 60 milioni d’anni fa e ci è rimasta. Questi elementi controllano una parte della risposta antivirale innata e hanno contribuito allo sviluppo di centinaia di motivi di DNA capaci di attivare i geni vicini in risposta all’interferone gamma, una proteina che interviene nel controllo delle cellule del sistema immunitario.
In modo simile, il gruppo di Joanna Wysocka, all’università di Stanford, ha scoperto che un sottogruppo di sequenze derivato da retrovirus endogeni HERVK, che esiste solamente nei primati ominoidei, esseri umani compresi, controlla sulla lunga distanza cromosomica l’attività di centinaia di geni attivi nelle cellule embrionali.
I motivi a dita di zinco sono indispensabili.
Il legame tra elementi trasponibili e genoma è rafforzato, nei vertebrati, da una famiglia di proteine dette a dita di zinco con un dominio KRAB (KZFP). Ogni proteina riconosce un unico motivo di DNA e induce un cambiamento della cromatina che lo circonda, reprimendo i geni che essa contiene. Ora, una parte dei KZFB riconosce direttamente gli elementi trasponibili. Di conseguenza, la proliferazione di questi trasposoni è contenuta e l’espressione dei geni circostanti regolata.
Didier Trono, della scuola politecnica di Losanna, ha proposto recentemente che le proteine KZFP controllano l’espressione dei geni importanti per lo sviluppo embrionale o neurale. Occasionalmente, sono sequestrati per reprimere una famiglia particolare di elementi trasponibili, in questo tessuto. Questi trasposoni si moltiplicano e si disperdono nel genoma, apportando dei nuovi siti di legame di KZFP vicino a nuovi geni. A lungo termine il processo si spegne, perché da una parte è bloccata dai KZFP e dall’altra, ognuna delle coppie accumula mutazioni che l’inattivano. Nonostante ciò, i motivi di legame di KZFP sono stati disseminati e sono attualmente in grado di regolare nuovi geni. Se le innovazioni così acquisite apportano un qualche avvantaggio selettivo, l’elemento trasponibile che trasporta il motivo sarà progressivamente selezionato, o fissato nella popolazione.
Insomma, la relazione tra i trasposoni e il genoma contribuisce ampiamente all’evoluzione della rete di regolazione genica. Gli elementi trasponibili apportano una plasticità inaudita nei genomi degli esseri viventi, ben al di là delle semplici mutazioni di DNA. Uno degli esempi più evidenti è la comparsa dell’immunità acquisita nei vertebrati, fondata sulla diversificazione degli anticorpi. Come David Schatz, all’università di Yale ha dimostrato, gli enzimi che iniziano queste reazioni, RAG1 e RAG2 derivano da un trasposone a DNA, noto come protoRAG. Anche il nostro sistema immunitario è il frutto dell’addomesticamento di un elemento trasponibile.
E la placenta fu…
Più recentemente nella storia evolutiva, l’evoluzione della placenta nei mammiferi (detti placentali) è legata all’infezione delle cellule sessuali o embrionali da parte di retrovirus. Tutti i retrovirus possiedono sulla superficie delle proteine dell’involucro, che rendono possibile la fusione delle membrane cellulari e virali, la prima fase dell’infezione. Queste proteine, talvolta, influiscono sull’azione del sistema immunitario. Ed anche se i retrovirus non sono funzionali dal punto di vista replicativo ed infettivo, i geni delle proteine dell’involucro, le sincitine, sono sempre funzionali e hanno un ruolo fisiologico essenziale.
Le sincitine, espresse nella superficie delle cellule del trofoblasto, portano alla fusione di queste cellule, una tappa chiave della placentazione. Esse contribuiscono anche alla tolleranza immunologica tra madre e figlio, evitando il rigetto. Inoltre, il gruppo di Thierry Heidmann, dell’Istituto Gustave-Roussy, ha dimostrato che l’addomesticazione degli involucri dei retrovirus endogeni ha avuto luogo numerose volte in maniera indipendente nel corso dell’evoluzione dei mammiferi.
Infine, un ultimo esempio, il gene Arc, implicato nella plasticità sinaptica, deriva da un retrotrasposone che è inserito nel genoma dell’antenato comune dei vertebrati terrestri, i tetrapodi.
Conclusioni
In conclusione, oltre al loro ruolo centrale nell’espressione genica, gli elementi trasponibili hanno contribuito all’emergere di funzioni fisiologiche essenziali come l’immunità, la riproduzione ed il funzionamento cerebrale.
In quanto costituiscono più della metà del nostro genoma, anche se una buona parte è stata addomesticato, è molto probabile che verranno scoperti molti altri esempi.
La nostra autostima dovrebbe soffrirne, dobbiamo molto a dei geni spesso definiti egoisti.
Federica Angius
https://www.microbiologiaitalia.it/virologia/gli-architetti-del-genoma-arrivano-dal-passato-linfluenza-dei-trasposoni/
mercoledì 5 giugno 2024
Gli scienziati trovano il genoma più grande di sempre. - Lucia Petrone
La scoperta.
Una piccola felce apparentemente insignificante che cresce solo su una remota isola del Pacifico è stata incoronata venerdì detentrice del Guinness World Record per avere il genoma più grande di qualsiasi organismo sulla Terra. La felce della Nuova Caledonia, Tmesipteris oblanceolata , ha più di 50 volte più DNA racchiuso nel nucleo delle sue cellule rispetto agli esseri umani. Se il DNA di una delle cellule della felce – che sono larghe solo una frazione di millimetro – venisse svelato, si estenderebbe fino a 106 metri hanno detto gli scienziati in un nuovo studio. In piedi, il DNA sarebbe più alto della torre che ospita la famosa campana del Big Ben di Londra.Il genoma della felce pesava ben 160 coppie di gigabasi (Gbp), la misura della lunghezza del DNA. Si tratta del 7% in più rispetto alla precedente detentrice del record, la pianta da fiore giapponese Paris japonica. Il genoma umano è relativamente esiguo: 3,1 Gbp. Se il nostro DNA venisse svelato, sarebbe lungo circa due metri. La coautrice dello studio Ilia Leitch, ricercatrice presso i Royal Botanic Gardens di Kew nel Regno Unito, ha dichiarato all’AFP che il team è stato “davvero sorpreso di trovare qualcosa di ancora più grande della Paris japonica “. “Pensavamo di aver già raggiunto il limite biologico. Stiamo davvero spingendo agli estremi della biologia”, ha detto. La felce, alta dai 5 ai 10 centimetri, si trova solo in Nuova Caledonia, un territorio francese del Pacifico recentemente teatro di disordini. Due membri del gruppo di ricerca si sono recati sull’isola principale, Grand Terre, nel 2023 e hanno lavorato con scienziati locali per lo studio, che è stato pubblicato. Il Guinness World Records ha assegnato alla felce l’ambito “titolo del genoma più grande”. La vittoria di “questa felce dall’aspetto innocuo” dimostra che “i detentori del record non sono sempre i più appariscenti all’esterno”, ha detto Adam Millward, caporedattore del Guinness World Records. Cos’è ancora un genoma? Si stima che gli esseri umani abbiano più di 30 trilioni di cellule nel nostro corpo. All’interno di ciascuna di queste cellule c’è un nucleo che contiene il DNA, che è come un “libro di istruzioni che spiega a un organismo come noi come vivere e sopravvivere”, ha spiegato Leitch. Tutto il DNA di un organismo è chiamato genoma. Finora gli scienziati hanno stimato la dimensione del genoma di circa 20.000 organismi, solo una frazione della vita sulla Terra. Tra gli animali il più grande è il dipnoo marmorizzato, con 130 Gbp. Sebbene le piante abbiano i genomi più grandi, possono anche averne di incredibilmente piccoli. Il genoma della carnivora Genlisea aurea è di soli 0,06 Gbp. Ma noi esseri umani non dobbiamo sentirci inadeguati quando ci confrontiamo con il potente T. oblanceolata . Tutte le prove suggeriscono che avere un genoma enorme è uno svantaggio, ha detto Leitch. Più DNA hai, più grandi devono essere le tue cellule per comprimerlo tutto.
domenica 13 agosto 2023
Progettati batteri in grado di rilevare il Dna del tumore: lo studio.
Sono come 'guardiani' invisibili, in grado di intercettare il nemico in maniera efficiente e veloce. A puntare su di loro è un gruppo di scienziati Usa, che ha ingegnerizzato dei batteri in grado di rilevare la presenza di Dna tumorale in un organismo vivo. Un'innovazione testata con risultati positivi nei topi che potrebbe aprire la strada a nuovi biosensori hi-tech capaci di identificare infezioni, tumori e altre malattie. I ricercatori dell'University of California San Diego e un gruppo di colleghi in Australia hanno descritto il passo avanti su 'Science'. Batteri come questi, in precedenza, erano stati progettati per svolgere varie funzioni diagnostiche e terapeutiche, ma non avevano la capacità di identificare specifiche sequenze di Dna e mutazioni al di fuori delle cellule. Il nuovo progetto - battezzato 'Catch' - nasce per fare proprio questo.
"Quando abbiamo iniziato 4 anni fa, non eravamo nemmeno sicuri che fosse possibile utilizzare i batteri come sensore per il Dna dei mammiferi", spiega il leader del team scientifico Jeff Hasty, professore della UC San Diego School of Biological Sciences e della Jacobs School of Engineering. "L'individuazione di tumori gastrointestinali e lesioni precancerose è un'interessante opportunità clinica a cui applicare questa invenzione". È noto che i tumori disperdono il loro Dna negli ambienti che li circondano. Molte tecnologie possono analizzare il Dna purificato in laboratorio, ma non sono in grado di rilevarlo lì dove viene rilasciato. I ricercatori hanno progettato e testato dei batteri con questa missione, utilizzando la tecnologia Crispr, dell'editing genetico.
"Molti batteri possono assorbire il Dna dal loro ambiente, un'abilità nota come competenza naturale", ha affermato Rob Cooper, co-autore dello studio, del Synthetic Biology Institute della UC San Diego. Hasty, Cooper e il medico australiano Dan Worthley hanno collaborato a un'applicazione di questa idea al cancro del colon-retto. Hanno iniziato a formulare la possibilità di ingegnerizzare i batteri che sono già prevalenti nel colon, come nuovi biosensori che potrebbero essere distribuiti all'interno dell'intestino per rilevare il Dna rilasciato da questo tumore.
Il team statunitense-australiano si è concentrato sull'Acinetobacter baylyi - un candidato con le qualità giuste - ingegnerizzandolo e testandolo come sensore per identificare il Dna di KRAS, un gene che è mutato in molti tipi di cancro e per discriminare tra la versione mutata e quella normale. "È stato incredibile quando ho visto al microscopio i batteri che avevano assorbito il Dna del tumore. I topi con tumori avevano sviluppato colonie batteriche verdi che avevano acquisito la capacità di crescere su piastre antibiotiche", ha affermato Wright.
I ricercatori stanno ora adattando la loro strategia di biosensori batterici a nuovi circuiti e diversi tipi di batteri per rilevare e trattare tumori e infezioni umane. In futuro, riflette Siddhartha Mukherjee, professore associato della Columbia University, non coinvolto nello studio, "le malattie saranno curate e prevenute da cellule, non da pillole. Un batterio vivente in grado di rilevare il Dna nell'intestino offre un'enorme opportunità" di schierare "una sentinella per cercare e distruggere il cancro gastrointestinale e molti altri".
La nuova invenzione richiede un ulteriore sviluppo e perfezionamento, precisano gli scienziati. Il team dell'UC San Diego sta continuando a ottimizzare questa strategia avanzata dei biosensori. "C'è un futuro in cui nessuno dovrà morire di cancro del colon-retto", auspica Worthley. "Speriamo che questo lavoro sia utile a bioingegneri, scienziati, e in futuro ai medici, nel perseguimento dell'obiettivo".
domenica 30 luglio 2023
Machu Picchu: l’analisi del DNA getta nuova luce sul popolo Incas. - Arianna Guastella
Una nuova analisi del DNA dei resti umani sepolti a Machu Picchu fornisce informazioni sui servitori degli Incas.
Gli uomini e le donne che servivano i reali Inca a Machu Picchu non erano locali; provenivano da terre lontane conquistate dall’impero, rileva un nuovo studio.
Un team internazionale di ricercatori ha analizzato l’antico DNA di oltre 30 persone sepolte a Machu Picchu, probabilmente servitori dell’élite Inca, e ha confrontato i dati genetici con il DNA di altri antichi resti umani e di persone moderne della regione.
I risultati hanno rivelato che i servi provenivano da tutti gli altopiani andini, così come da tutta la costa del Perù, secondo lo studio, pubblicato sulla rivista Science Advances.
Chi viveva a Machu Picchu?
Gli Incas governarono la regione andina del Sud America dall’inizio del XV secolo alla metà del XVI secolo, quando gli spagnoli presero possesso dell’impero. Più di un secolo prima dell’invasione spagnola, gli Inca costruirono un imponente palazzo sulle montagne del sud del Perù, probabilmente per l’imperatore inca Pachacuti, che regnò dal 1438 al 1471. Ma poco si sa sulle origini e sulla vita dei servi che gestivano la tenuta di Machu Picchu.
Circa 750 persone vivevano a Machu Picchu – tra cui l’imperatore, altri membri della famiglia reale Inca, ospiti e servitori permanenti – durante l’alta stagione tra maggio e ottobre, secondo lo studio. Molti reali erano serviti da uomini conosciuti come “yanacona”, che non erano Inca. Piuttosto, venivano spesso prelevati dalle terre conquistate e presentati in dono all’imperatore. Anche le donne conosciute come “aclla” furono prese dalle loro terre d’origine e date in moglie a questi servi maschi. Insieme, la yanacona e l’aclla provvedevano ai bisogni dell’imperatore e dei suoi ospiti mentre si dedicavano a banchetti, canti, danze e cacce e svolgevano importanti cerimonie religiose.
Nell’ultimo secolo di lavori archeologici a Machu Picchu, i ricercatori hanno scoperto le tombe di quasi 200 persone morte tra il 1420 e il 1532. Date le ceramiche semplici e non in stile inca sepolte con gli individui, si è a lungo ipotizzato che queste grotte funerarie contenevano i resti dei servi yanacona e aclla che frequentavano la famiglia reale. Ricerche precedenti che utilizzavano analisi biochimiche suggerivano inoltre un alto livello di diversità etnica tra la popolazione sepolcrale di Machu Picchu.
Per verificare ulteriormente l’ipotesi che le persone sepolte fossero servitori portati lì da diverse parti del Sud America, i ricercatori hanno analizzato i dati del DNA antico di 34 persone trovate nei quattro cimiteri di Machu Picchu, così come il DNA di 36 persone moderne e antiche della Valle di Urubamba, chiamata anche Valle Sacra, a nord della capitale Inca di Cusco.
I risultati hanno rivelato che “Machu Picchu era sostanzialmente più diversificato geneticamente rispetto ai villaggi rurali contemporanei nelle Ande”, ha dichiarato Lucy Salazar, un’archeologa dell’Università di Yale che ha condotto lo studio.
Inoltre, il team ha trovato una differenza significativa tra gli antenati genetici dei servitori maschi e femmine: la maggior parte degli individui maschi proveniva dalle regioni degli altipiani, mentre le femmine avevano antenati molto più diversi e non degli altipiani.
Nel testare gli scheletri per la parentela biologica, i ricercatori hanno trovato solo una coppia di parenti di primo grado: una madre e una figlia sepolte l’una vicino all’altra. La madre sembra provenire dalle pianure amazzoniche, mentre la figlia è cresciuta negli altopiani o nelle Ande costiere. La mancanza di ulteriori relazioni biologiche suggerisce che i servi arrivarono a Machu Picchu come individui piuttosto che come comunità o famiglie allargate, hanno concluso i ricercatori.
Ken-ichi Shinoda, antropologo e direttore del Museo nazionale della natura e della scienza del Giappone, non coinvolto nello studio, ha dichiarato che “considerando che Machu Picchu era una città significativa all’epoca, non sorprende che persone provenienti da varie regioni andine si siano riunite qui”. Shinoda e il suo team hanno precedentemente analizzato il DNA di scheletri in luoghi di sepoltura non d’élite intorno a Machu Picchu e hanno trovato una diversità genetica molto inferiore.
Gli scheletri nel nuovo studio, che sono stati scavati e portati alla Yale University nel 1912, sono stati oggetto di richieste di rimpatrio fino a quando non sono stati tutti restituiti al Perù nel 2012. In passato, “non potevo analizzarli”, ha detto Shinoda. “Ora che è diventato possibile, sono lieto che siano state fatte nuove scoperte”.
Mentre le nuove analisi rivelano informazioni sulle origini e sulla vita dei servitori che gestivano Machu Picchu, rimangono interrogativi sulla vita dei reali.
“Nonostante i limiti intrinseci”, hanno scritto i ricercatori, “le nostre analisi degli individui non elitari dimostrano che le informazioni genomiche, in combinazione con fonti archeologiche ed etnostoriche, possono rivelare una visione più sfumata e completa della vita quotidiana a Machu Picchu rispetto a quella disponibile in passato”.
https://reccom.org/machu-picchu-dna-getta-nuova-luce-sul-popolo-incas/
giovedì 1 dicembre 2022
Trovata la chiave dell’intelligenza dei polpi, è la stessa degli esseri umani.

È nel vasto repertorio di piccole molecole di Rna nel tessuto nervoso.
La scoperta, pubblicata sulla rivista Science Advances e guidata dal Centro tedesco Max Delbrück, rende questi molluschi un’eccezione unica tra gli animali invertebrati, che possono ricordare informazioni, riconoscere le persone e forse perfino sognare.
I polpi in particolare, dal punto di vista evolutivo, rappresentano un caso unico: possiedono sia un grande cervello centrale, sia un sistema nervoso periferico, che è in grado di agire in maniera indipendente.
sabato 2 aprile 2022
Completata la mappa del genoma umano, Dna senza segreti. - Enrica Battifoglia
Ci sono voluti 21 anni e tecnologie avanzatissime, ma finalmente il Dna umano non ha più segreti e diventa più facile fare passi in avanti nella medicina personalizzata, con la diagnosi di malattie finora impossibili da riconoscere, nella genetica delle popolazioni e nella possibilità di riscrivere il Dna.
I risultati sono pubblicati in sei articoli in un numero speciale della rivista Science, che a questo traguardo dedica la copertina.
"E' come avere un vocabolario" del Dna, osserva il genetista Giuseppe Novelli, dell'università di Roma Tor Vergata. "Abbiamo dei termini di riferimento che finalmente rendono possibile fare la diagnosi di alcune malattie" rare, caratterizzate da sequenze genetiche instabili.
"Non basta sequenziare il Dna: bisogna saperlo leggere e bisogna interpretarlo. In caso contrario, risulta molto difficile fare la diagnosi malattie dovute a sequenze ripetute, con interruzioni che in passato non si potevano vedere".
Il grande libro della vita era stato tradotto per la prima volta nel 2001, ma i computer di allora non erano riusciti a decifrare tutti i passaggi e avevano lasciato degli spazi bianchi, che complessivamente corrispondevano all'8% del genoma. Solo adesso queste lacune sono state colmate e diventa possibile leggere il Dna umano dall'inizio alla fine senza interruzioni, grazie al lavoro fatto dal consorzio internazionale chiamato Telomere-to-Telomere (T2T). "Stiamo vedendo capitoli che non sono mai stati letti prima", scrivono i ricercatori. I nuovi capitoli corrispondono a 200 milioni di lettere, che complessivamente equivalgono all'informazione contenuta in un cromosoma. Era come avere una mappa di new York senza Manhattan, dicono i ricercatori.
"Se ottenere il sequenziamento del Dna è come mettere insieme un puzzle, il genoma di riferimento è avere l'immagine del puzzle finito sulla scatola: ti aiuta a mettere insieme i pezzi", ha detto uno degli autori della ricerca, l'ingegnere biomedico del National Institute of Standards and Technology (Nist), Justin Zook. Le parti mancanti comprendono sequenze che si ripetono molte volte e ora è chiaro che proprio nelle ripetizioni si nasconde il segreto della diversità umana, osserva la genetista Rachel O'Neill, dell'Università americana del Connecticut e responsabile scientifica del progetto T2T. Per portare alla luce questo lato ancora nascosto del Dna umano "ci sono voluti nuovi metodi di sequenziamento del Dna e di analisi computazionale", rileva il genetista Francis Collins, consulente scientifico della Casa Bianca ed ex direttore del National Institutes of Health (Nih). "E' valsa la pena aspettare", aggiunge, perché "adesso emerge una varietà di sorprendenti caratteristiche architettoniche, con importanti conseguenze per la comprensione dell'evoluzione umana, della variazione e della funzione biologica". Il nuovo libro del Dna è anche a prova d'errore, considerando che i ricercatori hanno utilizzato una sorta di correttore automatico, un programma chiamato Merfin che analizza le sequenze e corregge gli eventuali errori.
lunedì 27 settembre 2021
Ricostruito il Dna degli Etruschi. - Paola Catani
Ricostruito il Dna degli Etruschi (fonti: in primo piano particolare di un affresco etrusco del 510 a.C, diDave & Margie Hill/Kleerup; sullo sfondo la doppia elica del Dna, di Christoph Bock, Max Planck Institute
Erano cugini degli Italici, ma la loro lingua resta un mistero.
Etruschi cugini degli Italici, lo svela il Dna antico, ma la lingua parlata da questo popolo rimane un mistero. A provare la stretta parentela uno studio genomico definito il più grande mai realizzato sugli Etruschi, condotto da un team di studiosi internazionali, coordinato da Cosimo Posth, del dipartimento di Archeogenetica dell'Istituto tedesco Max Planck per la storia delle Scienze umane a Jena e condotto con le università di Firenze, Tubinga e Jena. Per l'Italia hanno contribuito anche le università di Siena, Napoli Federico II, Ferrara e Padova. Pubblicata sulla rivista Science Advances, la ricerca ha esaminato il Dna di 82 individui vissuti in Italia nell'arco di quasi 2000 anni, dall'800 a.C. al 1.000 d.C, in dodici siti, tra Toscana e Alto Lazio.
I risultati? Gli Etruschi "condividono il profilo genetico dei Latini della vicina Roma e gran parte del loro genoma derivi da antenati provenienti dalla steppa Eurasiatica durante l'età del bronzo". Lo studio, a cui hanno preso parte ricercatori degli Atenei di Firenze, Siena, Ferrara e del Museo della Civiltà di Roma, di Germania, Stati Uniti, Danimarca e Regno Unito, risolve così, si spiega, "l'enigma sulle origini di questa cultura altamente avanzata e ancora poco conosciuta", fiorita durante l'età del ferro nell'Italia centrale, e che ha incuriosito gli studiosi per millenni, coivolgendo storici illustri già dai tempi del greco Erodoto.
Per quest'ultimo discendevano da gruppi migratori anatolici o egei. Per gli archeologici invece hanno avuto un'origine locale, ipotesi suffragata in passato da alcune ricerche su Dna antico. E ora confermata da questa ricerca che fornisce "risposte definitive" sulle origini degli Etruschi. Resta però il mistero della loro lingua, non indoeuropea, estinta, solo in parte compresa. Se "i gruppi legati alla steppa Eurasiatica furono probabilmente responsabili della diffusione delle lingue indoeuropee, ora parlate in tutto il mondo da milioni di persone, la persistenza di una lingua etrusca non indoeuropea in Etruria è un fenomeno intrigante - si spiega - che richiederà un'ulteriore indagine".
"Questa persistenza linguistica, combinata con un ricambio genetico, sfida la tesi che i geni siano uguali alle lingue - afferma David Caramelli, docente di antropologia all'Università di Firenze - e suggerisce uno scenario più complesso che potrebbe aver coinvolto l'assimilazione dei primi popoli italici da parte della comunità linguistica etrusca, forse durante un periodo prolungato di mescolanza nel secondo millennio a.C."
Lo studio ha anche rivelato "importanti trasformazioni genetiche associate a successivi eventi storici" con riferimento sempre all'Italia centrale: una, durante il periodo imperiale romano, legata alla commistione con le popolazioni del Mediterraneo orientale che probabilmente includevano schiavi e soldati trasferiti attraverso l'Impero Romano; l'altra nell'Alto medioevo, identificata con la diffusione di antenati dell'Europa settentrionale nella penisola in seguito al crollo dell'Impero romano d'Occidente.
"Questo cambiamento genetico - afferma Johannes Krause, direttore del Max Planck Institute per l'evoluzione antropologica - descrive chiaramente il ruolo dell'Impero Romano nello spostamento delle persone su larga scala in un momento di maggiore mobilità socioeconomica e geografica". "L'Impero Romano - afferma Cosimo Posth, docente all'Università di Tubinga e Centro Senckenberg per l'evoluzione umana e il paleoambiente - sembra aver lasciato un contributo duraturo al profilo genetico degli europei meridionali, colmando il divario tra le popolazioni europee e del Mediterraneo orientale sulla mappa genetica dell'Eurasia occidentale"
ANSA
sabato 7 marzo 2020
Taglia-incolla Dna usato per la prima volta nel corpo umano.
La Crispr-Cas applicata per la prima volta in vivo nelle cellule della retina (fonte: P. Motta/Università Sapienza di Roma/SPL)
Per correggere le cellule malate nell'occhio e non in provetta.
mercoledì 30 ottobre 2019
Questa immagine, chiamata Foto 51, è considerata la più importante fotografia di tutti i tempi. Ecco la sua storia. Avvincente. - Mariella Bussolati

La Foto 51, fatta da Raymond Gosling sotto la supervisione di Rosalind Franklin. Wikipedia.
Foto 51, chiamata così perché era la cinquantunesima che i suoi autori avevano ottenuto, è un’immagine di diffrazione a raggi X di un filamento della proteina genica da cui dipende la trasmissione delle informazioni che controllano lo sviluppo di ogni organismo, il Dna. Venne catturata da Raymond Gosling, uno studente, ma la sua paternità va attribuita alla scienziata con cui lavorava, Rosalind Franklin, una biochimica inglese esperta in cristallografia e una campionessa assoluta di indagini a raggi x su varie sostanze. E’ grazie a quelle strisce che si è potuto capire che il Dna era fatto di due molecole intrecciate tra loro, una doppia elica.

- Rosalind Franklin. National Portrait Gallery

- Watson, Crik e il modellino del Dna a doppia elica.






